K2) Amylas Ep. 1
K2) Laboration
Nivå: A, APA 7, hög nivå.
Jag har genomfört en laboration som egentligen inte har något förutbestämt syfte än att undersöka hur pH som faktor påverkar enzymaktiviteten hos amylas.
Metod
Jag bespar er tid eftersom denna laboration är välkänd och skriver därför mycket kort.
Amylas blandad med stärkelse och buffer tillsattes med jämna mellanrum till en droppe Lugol's lösning fram till att inget utslag längre observerades, dvs. droppen skiftade inte färg från ursprunget orange till något annat. För att mäta detta togs tid från att första droppen skiftat färg till den sista som inte gjorde det.
Fråga 1:
Vad hade en passande frågeställning och syfte varit?
pH 3, 4, 5, 6, 7, 8, 9, 10 och 11 testades. Under laborationen landade experimentet i att jag försökte hitta vilket pH som enzymaktiviteten var som störst genom reaktionshastighet.
Hade ett bra syfte varit "att undersöka vid vilket pH som enzymaktiviteten är som störst utifrån reaktionshastighet"? Kom gärna med idéer och förslag. Utvecklar och förfinar gärna syftet. Kändes som att jag sökte ett optimum snarare än "vid vilka pH slutar enzymet fungera".
Fråga 2:
Hur hade resultatet kunnat presenterats på bästa möjliga sätt?
För pH 3 och 11 kunde ingen sluttid mätas eftersom det för pH 3 och 11 verkar har denaturerats.
För pH 10 slutade jag mäta tiden och undersöka färgskiftningen eftersom den gick väldigt långsamt och efter 12 minuter knappt hade ändrat färg.
Min tanke är därför att det inte går att sammanställa i ett linjediagram där hastigheten ställs mot pH eftersom det för pH 3, 10 och 11 inte finns några definitiva värden.
Ligger presentation genom tabell som bästa och enda alternativ eller finns det något sätt att presentera öppna värden (typ >12 min) i ett linjediagram?
Detta var mina första frågor angående laborationen.
Uppskattar all hjälp otroligt mycket, tveka inte att ställa frågor om något steg. Jag ville hålla mig kortfattad i inledningen för att inte frågan ska bli svårläst!
Tack på förhand.
Mesopotamia skrev:K2) Laboration
Nivå: A, APA 7, hög nivå.
Jag har genomfört en laboration som egentligen inte har något förutbestämt syfte än att undersöka hur pH som faktor påverkar enzymaktiviteten hos amylas.
Att undersöka effekten av pH på amylas, kan direkt syfta till att undersöka/lära sig om/hur pH påverkar enzymer.
Våra celler består ju till största delen av vatten, och resten av de ämnen vi består har egenskaper som gör att de fungerar bra tillsammans i vatten. Ämnena innehåller många (olika) funktionella grupper som interagerar med vatten och varandra (hydroxi-, karbonyl-, amin- amid-, estrar- o.s.v.) och de funktionella gruppernas egenskaper påverkas av om de är i syra- eller basformen. Och som du kanske redan tänker nu, på så vis kommer pH in!
Gruppernas egenskaper påverkar även de större molekyler som de finns i t.ex. enzymer, för om pH ändras ett par enheter (7 till 9) förändras protoneringen på massvis med funktionella grupper, och de kan få en annan laddning, och binda/interagera med sin omgivning på ett lite annorlunda sätt. Men "omgivningen" (del av proteinet) är optimerad för att se ut på precis rätt sätt, ändras det för mycket ändras strukturen och den optimerade omgivningen finns inte längre.
Metod
Jag bespar er tid eftersom denna laboration är välkänd och skriver därför mycket kort.
Amylas blandad med stärkelse och buffer tillsattes med jämna mellanrum till en droppe Lugol's lösning fram till att inget utslag längre observerades, dvs. droppen skiftade inte färg från ursprunget orange till något annat. För att mäta detta togs tid från att första droppen skiftat färg till den sista som inte gjorde det.
Fråga 1:
Vad hade en passande frågeställning och syfte varit?
pH 3, 4, 5, 6, 7, 8, 9, 10 och 11 testades. Under laborationen landade experimentet i att jag försökte hitta vilket pH som enzymaktiviteten var som störst genom reaktionshastighet.
Hade ett bra syfte varit "att undersöka vid vilket pH som enzymaktiviteten är som störst utifrån reaktionshastighet"? Kom gärna med idéer och förslag. Utvecklar och förfinar gärna syftet. Kändes som att jag sökte ett optimum snarare än "vid vilka pH slutar enzymet fungera".
Varför inte kombinera båda? Dels kan syftet vara att få reda på vid precis vilket pH som aktiviteten är högst, och inom vilket pH-intervall som enzymet är aktivt (utanför intervallet "slutar enzymet fungera").
Fråga 2:
Hur hade resultatet kunnat presenterats på bästa möjliga sätt?
För pH 3 och 11 kunde ingen sluttid mätas eftersom det för pH 3 och 11 verkar har denaturerats.
För pH 10 slutade jag mäta tiden och undersöka färgskiftningen eftersom den gick väldigt långsamt och efter 12 minuter knappt hade ändrat färg.
Min tanke är därför att det inte går att sammanställa i ett linjediagram där hastigheten ställs mot pH eftersom det för pH 3, 10 och 11 inte finns några definitiva värden.
Ligger presentation genom tabell som bästa och enda alternativ eller finns det något sätt att presentera öppna värden (typ >12 min) i ett linjediagram?
Om du skapar ett diagram med aktivitet/hastighet mot pH, så brukar det gå att se en trend. Och ser du ingen aktivitet vid pH 3, så är antingen något knas, eller så är hastigheten faktiskt noll för att proteinet denaturerats...
Detta var mina första frågor angående laborationen.
Uppskattar all hjälp otroligt mycket, tveka inte att ställa frågor om något steg. Jag ville hålla mig kortfattad i inledningen för att inte frågan ska bli svårläst!
Tack på förhand.
Tack så mycket för ditt suveräna svar mag1! Jag ska läsa igenom det ett par gånger till och återkommer om jag fundera över något mer.
Men hur blir det med de öppna värdena i linjediagramet, typ >12 min? Jag har liksom inget definitivt uppmätt värde för pH 10...? Kan man visa det på något sätt visuellt i diagrammet? Har tyvärr aldrig nämnts av vår lärare.
Ofta brukar pH-aktivitetskurvor visas som en kurva, som sammanbinder datapunkterna. Kurvan brukar vara klockformad (lite bredare än en Gaussisk distribution). Och plottar du de datapunkter du har styr de hur kurvan ser ut, saknas t.ex. ett värde för pH 10 får du lämna den cellen i excel tom, och anpassa kurvan mellan pH 9-11, motsvarande sammanbinda de punkterna. Det går att göra på papper också, med en linje mellan dem. Snyggast brukar det bli om du anpassar kurvan efter trenden som finns innan. T.ex. faller aktiviteten mellan pH 8-9 och är noll vid pH 12 - kan du anpassa kurvan efter den fallande trenden.
Du kommer bara ha mätdata på diskreta punkter, d.v.s. de pH du mätt vid. Och mellan dessa punkter vet du inte hur aktiviteten är, men du kan nog med motivering göra antagandet att aktivitetsförändringen fortsätter. Hur mycket du behöver motivera beror på hur mätvärdena är.
Och igen, dina data är för diskreta pH, linjen mellan punkterna i ett sådant diagram är illustrativa och visar trenden. Är mätvärdena normalfördelade kommer kurvan bli Gaussisk, men är mätvärdena annorlunda kommer ju även kurvans form annorlunda - vilket i sig innehåller en hel del information att tolka (mätfel eller biokemi etcetera).
Du har inte angivit hur era data skall presenteras, men en tabell är nog enklast. Kurvan är dock mer illustrativ, och belyser en central del av enzymers kinetik (så jag skulle säga att den är väl värd att göra bara, för att illustrera hur enzymer påverkas och fungerar).
Prova att göra kurvan och återkom om du fastnar eller vill diskutera mera.
Hej mag1!
Nu har jag gjort kurvan och anpassat datapunkterna utan värde efter trenden:

Det finns inget krav på typ av presentation. Det känns som att det kommer krävas mer motivering om kurvan bifogas, särskilt för pH som hade öppna tidsvärden (minst > X min), än om det presenteras i en tabell, då man kan skriva just >10 min etc. Vad tycker du? Jag strävar efter högsta betyg och gör därför det som du tycker passar bäst.
(Formateringen och rubriker har jag inte finslipat ännu nu, fokuserar mer på datan.)
Tack för hjälpen!
Utifrån vad man skulle förvänta sig av amylas, och att det är data från första gången du gjort laborationen så ser kurvan bra ut, d.v.s. resultatet ser ut som man kan förvänta sig med alfa-amylas (som jag misstänker ni använt).
Det framgår i kurvan vid vilket pH intervall som aktiviteten är som högst, och det är tydligt att du inte kunde se någon aktivitet vid pH 3 eller 11 - och du har redan börjat resonera kring att det är kopplat till att enzymet är denaturerat. Du kan så klart även presentera tiderna i en tabell, och sedan bara skriva att datat även visas sammanställt i diagrammet. Det är ju samma data, men diagrammet illustrerar det lite tydligare - vilket gör det enklare att tolka och diskutera.
Skulle du haft fler mätpunkter och upprepat experimentet flera gånger, skulle nog kurvan (med medianvärdena från de upprepade experimenten plottade) vara lite mjukare och mer "snygga att titta på" - men trenden är tydlig.
Det var för att se trenden som jag föreslog att du skulle göra detta diagram. Den illustrerar att aktiviteten är beroende av pH, men aktiviteten försvinner inte direkt vid t.ex. pH 5 utan den börjar att minska redan mellan pH 6-5, men det finns lite aktivitet kvar mellan pH 5-3.
Gällande motivationen av "öppna tidsvärden", så är ca tider helt okej, det är den precision som du har. Och för de pH där det tog mer än 10 minuter, så kan du nämna just det - att t.ex. för pH 3 skedde ingen färgförändring efter maxtiden 10 minuter, och därför är den aktiviteten satt till noll (du såg ju ingen aktivitet alls där, så det är inte fel, men det är bra att nämna för rapportläsaren vad detta värde noll innebär).
Om du känner dig ambitiös, kan du söka fram inom vilket pH intervall som amylaset är aktivt. Ni använde kanske inköpt amylas från t.ex. gris, det kanske finns detaljer i laborationsinstruktionerna om vilket amylas det var? Med den informationen kan du kommentera på hur rimligt ditt resultat är, och du hittar kanske även lite information om detta enzym som kan vara intressant (både för din del och) för rapporten.
För all del!
Grymt!
Jag läser igenom ditt svar imorgon eftersom det börjar bli sent och har NP imorgon.
Återkommer då!
Hej mag1!
Jag har ytterligare en fråga angående detta.
Jag har svårt att kontrollera om det jag skrivit i mitt diagram stämmer med avseende på enhet.
Kan man säga att jag mätt reaktionshastigheten? Visst är enheten 1/s?
Eller är det enzymaktiviteten jag mätt? (Tvivlar på det.)
Tack för hjälpen!
Beror lite på vad 1/s innebär i detta sammanhang. Det ser ut som om du skapat denna enhet från tiden det tog tills dess att färgomslaget skedde.
Hastigheten du kan få är "substratet är nedbrutet efter X minuter", motsvarande "total substratomvandling efter X minuter", som blir ett mått på aktiviteten då du kan inte mäta mängden bildad produkt. Men det spelar ju ingen roll, för förbrukningen av substratet går lika bra att följa. Och det enda värdet du kan får är X minuter, d.v.s. tiden då substratet är nedbrutet (i alla fall så mycket av substratet att det inte kan detekteras genom bildande av polyjodidjonkomplexet).
Aktiviteten hos enzym brukar anges på mer komplicerade sätt (med olika modeller beroende på enzymets kinetik, men det är universitetskemin). Men tiden det tar att bryta ner en och samma mängd stärkelse innehåller även den informationen om hastigheten.
I och med att du inte söker en exakt hastighet utan vill jämföra relativa hastigheter vid olika pH, kan du godtyckligt sätta mängden substrat till 1 och helt enkelt dela den mängden med tiden det tar att omvandla allt substrat, med brasklappen att all tid över 12 minuter sätter du till 0, d.v.s. ingen aktivitet ses över huvud taget.
mag1 skrev:Hastigheten du kan få är "substratet är nedbrutet efter X minuter", motsvarande "total substratomvandling efter X minuter", som blir ett mått på aktiviteten då du kan inte mäta mängden bildad produkt. Men det spelar ju ingen roll, för förbrukningen av substratet går lika bra att följa. Och det enda värdet du kan får är X minuter, d.v.s. tiden då substratet är nedbrutet (i alla fall så mycket av substratet att det inte kan detekteras genom bildande av polyjodidjonkomplexet).
I och med att du inte söker en exakt hastighet utan vill jämföra relativa hastigheter vid olika pH, kan du godtyckligt sätta mängden substrat till 1 och helt enkelt dela den mängden med tiden det tar att omvandla allt substrat, med brasklappen att all tid över 12 minuter sätter du till 0, d.v.s. ingen aktivitet ses över huvud taget.
Super!
Jag tror att jag hänger med så långt; det är alltså "relative reaction rate". Visst?
Ja det går bra som engelsk term.
Jag har försökt förstå amylas uppbyggnad, men finner de olika termerna förvirrande.
Det jag hittar är att enzymet består av 3 domäner, ingen subenhet, dvs. en enda lång kedja.
Hur tolkar man de två bilderna nedan? På ena ser det ut som 3, på den andra som 4.
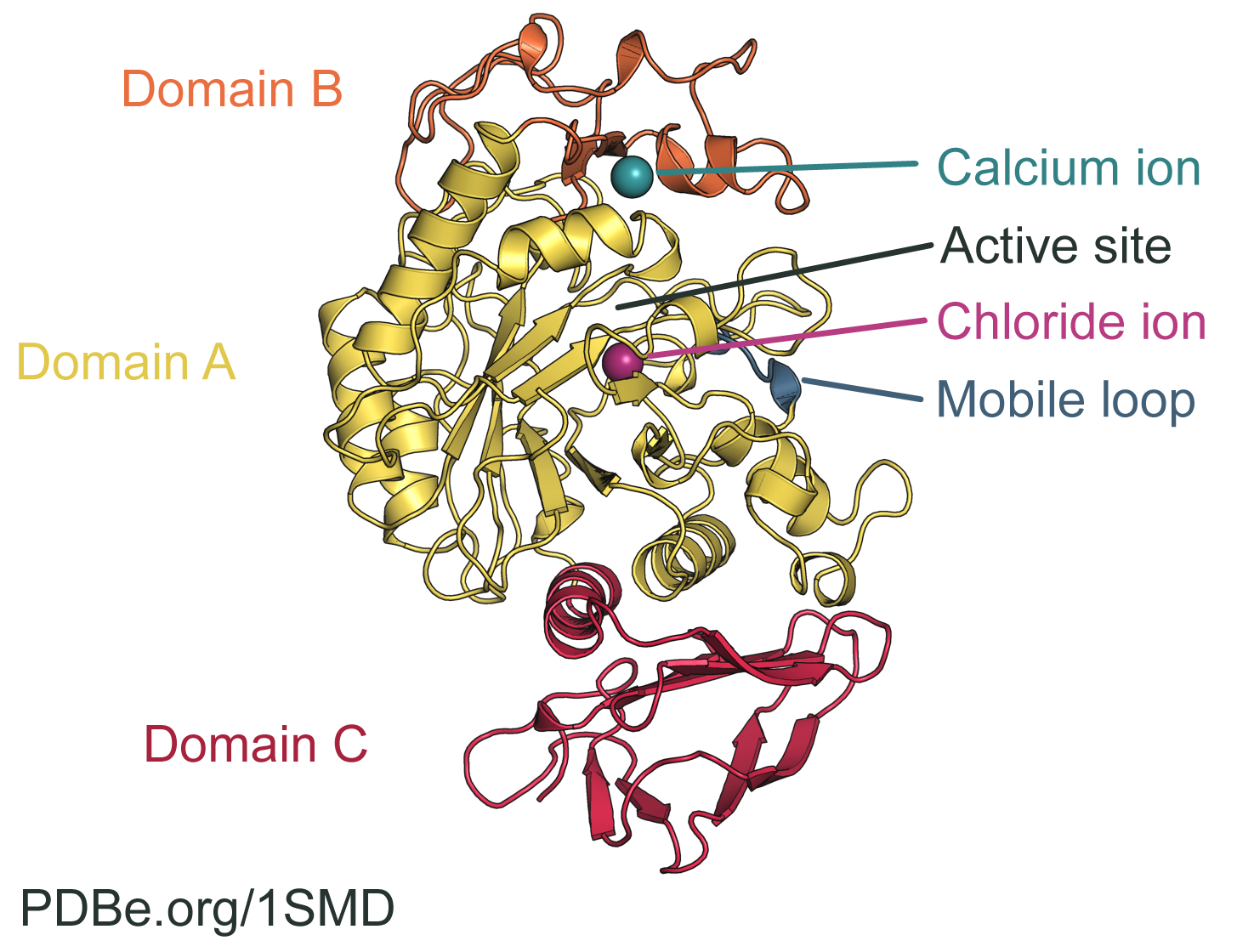

Eller är färgerna inget bra sätt att försöka lista ut domän och subenhet på?
Tillägg: 8 maj 2024 20:41
Säger man då att den inte har någon kvartärstruktur eftersom den består av en enda kedja?
Beskrivningen av ett proteins struktur kan göras på flera vis, i olika detaljnivå och oftast utifrån tidigare kända exempel. Det blir enklare att beskriva strukturens delar var och en för sig, om domäner/motiv redan setts i andra strukturer. Man kan då beskriva en struktur som bestående av motiv 1, 2, 3 o.s.v. Och sedan hur de olika motiven binder till varandra, och bildar tertiärstrukturen, och det kan kopplas till kvarärstrukturen (om det finns en sådan).
En liknelsen kan vara att en klädsel (proteinet) består av mössa, en vante, tröja, och kortbyxor (där plaggen är t.ex. domäner). Domänerna kan beskrivas var och en för sig som liknande alfa/beta-tunna (domän A). Med ett känt exempel på domäntypen kan man beskriva den övergripande strukturen av samma/liknande domänen i andra proteiner, som t.ex. alfa/beta-tunna. Beskrivningen blir då snarare "ser ut som alfa/beta-tunna", och sedan beskrivs hur just denna domän skiljer sig exempel-domänen. Motsvarande tröjan (alfa/beta-tunnan) är vävd av bomull och lite mindre än exempeltröjan, som var den första som beskrevs.
De olika motiven/domänerna återkommer i flera olika proteiner, evolutionen har använt samma domän i flera olika sammanhang/funktioner. Därför kan alfa/beta-tunnan finnas i allt från helt ensam till i kombination med flera andra domäner - det är dock fortfarande samma övergripande domänstruktur (en ylletröja är precis som en bomullströja fortfarande en tröja, även om de fyller olika funktion i just sitt protein).
Alfa-amylaset som du hittat figurer för finns som en ensam molekyl bestående av en enda polypeptidkedja, d.v.s. den saknar kvartärstruktur.
Färgerna illustrerar ganska tydligt vilka de olika domänerna är. Och samtliga domäner tillhör en och samma subenhet (då det endast är en polypeptidkedja).
Är du med på varför de olika jonerna behövs?
Måste bara säga att om du inte redan är lärare så har du otrolig talang!!! Dina förklaringar är alltid så genomtänkta och tydliga, tack innerligt.
Nu hänger jag med mer på domän, subenhet och de olika strukturerna.
Jonerna har jag läst mig till att de är aktivatorer som är viktiga för enzymets tertiära struktur, stabilitet och funktion. Stämmer det?
Tackar, tackar, det var fina ord! Glädjande att höra.
Och ja jonerna behövs som aktivatorer, och påverkar enzymets struktur, fast på lite olika vis.
Kalciumjonen på den stora skalan - när kalciumjonen binder in i amylaset, ändras strukturen av domänen B så att den ordnas mer, och får den strukturen som ses i figurerna i inlägg #12. Utan kalciumjonen saknas denna struktur hos domänen B, och den delen av proteinet är då istället så pass rörlig att substratet (stärkelsen) inte kan binda till den aktiva ytan - och då kan inte enzymet katalysera hydrolysen av stärkelsen (katalysatorn, d.v.s. den "optimalt aktiva strukturen" av amylaset existerar helt enkelt inte riktigt utan kalciumjonen). Kalciumjonen ser i princip till att forma en del av platsen där substratet skall bindas in i aktiva ytan.
Avstånd i strukturer brukar av tradition anges i enheten Å, Ångström(efter den svenske fysiken Anders Ångström), vilket är 10-10 meter. Och omorganisation/struktureringen av domänen B, som orsakas av kloridjonen, är i storleksordningen tiotals Å så relativt stor (proteinet är ca 70 Å i den längsta riktningen, upp-ner i din bild)
Kloridjonen påverkar i det (pytte)lilla. Den binder i närheten av den aktiva ytan, och inbindningen av kloridjonen ser till att aminosyraresterna som utför den kemiska reaktionen, hamnar optimalt i förhållande till varandra. Och de avstånd som dessa aminosyraresters sidokedjor rör sig är i storleksordningen 0.1 Å. Men genom att de avstånden förändras på det sättet i den aktiva ytan (som en konsekvens av att kloridjonen binder in) fås en optimal geometri i den aktiva ytan - så att aktiveringsenergin för hydrolysen av stärkelsen sänks så lågt att reaktionen kan ske t.ex. i våran mun/tarm.
Intressant! Är detta man djupdyker mer inom universitetskemin?
Ska läsa igenom svaret noggrant för att se till att jag förstår varje detalj och återkommer om jag har funderingar!
Mesopotamia skrev:Intressant! Är detta man djupdyker mer inom universitetskemin?
Inom vissa delar kan det dyka upp. Det är i princip det som kallas strukturbiologi, d.v.s. studier av proteiners strukturer och funktion, och det brukar dyka upp lite där och där under grundkurser. Men djup blir det inte förrns under påbyggnadskurser inom strukturbiologi, och enzymologi.
Hej mag1!
Laborationsarbetet fortsätter, jag håller på att fundera över min diskussion.
Här är vad jag kommit fram till:
1. Jag kommer diskutera dels noggrannheten och att man endast kan säga att det mest optimala ligger mellan två värden, och därmed inte veta exakt var det ligger.
2. Diskutera denaturering av enzymet och dess struktur. Här planerar jag att ta upp det du skrev om Ångström och redan små förändringar har stor påverkan på både aktiviteten och funktionen, det vill säga, om överflödet av vätejoner påverkar klorid och kalciumjonerna, för visst gör de det? Även hydroxidjonerna? Är det laddningen som påverkar att de förflyttar sig?
Jag har även klurat på om det kan vara så att de kan påverkar den aktiva siten, dvs. blockera den genom att binda till någon negativ / positiv laddning som finns där och hindra stärkelsen från att binda. Ligger det något i det?
För pH 3 och 11 förstörs enzymt totalt, men för lite högre pH där hastigheten var mycket lägre, har det med att substansmängden icke-denaturerade enzym har minskat rejält, eller att det finns någon som gör de långsammare? Jag lutar åt det första, för jag tänker mig att antingen fungerar enzymet eller så gör det inte. Korrekt? Själv är jag osäker.
3. Teori kopplad till just pH 6-7, det vill säga, exoenzymet anpassats efter förhållanden som bakterier trivs i, vilka ligger runt det pH:et, varför vet jag inte. Har det någon evolutionär orsak? Vi använde som du misstänkte bakteriellt amylas. De verkar inte kolonisera människor, så att salivens lägre pH verkar inte ha med det att göra som man annars kan tro för bakterier som lever i munhålan och använder amylas. Tillägg: kom just ihåg sura marker då bakterien lever i jord, kan det vara en evolutionär sak, alltså näringsfattig jord där även pH är lågt?
4. Felkällor kring okulär besiktning av färg, att det kanske ändå fanns viss mycket svag aktivitet runt pH 3 och 11 som inte kunda upptäckas, för visst kan man prata om aktivitet i detta sammanhang?
Ser framemot at höra vad du tycker!
Hälsningar.
🗹 Aktualiserar tråden.
Mesopotamia skrev:Hej mag1!
Laborationsarbetet fortsätter, jag håller på att fundera över min diskussion.
Här är vad jag kommit fram till:
1. Jag kommer diskutera dels noggrannheten och att man endast kan säga att det mest optimala ligger mellan två värden, och därmed inte veta exakt var det ligger.
Ja typ så, högst aktivitet såg du vid pH 6. Du kan även sätta detta intervall, mellan två pH värden, som en aktivitetsnivå - t.ex. högre än 30% av den maximala aktiviteten sågs inom pH 5-9, eller ännu smalare 50% av den maximala aktiviteten fanns mellan pH 5,5-7,5 i diagrammet. Då uttalar du dig inte om de data du saknar (t.ex. pH 5,5 och 6,5) utan om trenden i kurvan - som illustrerar hur pass pH känsligt enzymets aktivitet är.
2. Diskutera denaturering av enzymet och dess struktur. Här planerar jag att ta upp det du skrev om Ångström och redan små förändringar har stor påverkan på både aktiviteten och funktionen, det vill säga, om överflödet av vätejoner påverkar klorid och kalciumjonerna, för visst gör de det? Även hydroxidjonerna? Är det laddningen som påverkar att de förflyttar sig?
pH förändrar protoneringen/laddningen på alla sidokedjor - så på sätt och vis påverkas bindandet av jonerna av pH.
Samtidigt påverkas även proteinets struktur övergripande. Sekundärstrukturelementen (alfahelixar och betasträngar/flak) bildar just t.ex. en helix inom ett pH intervall, och utanför detta intervall "smälter" helixen och aminosyrorna blir mindre ordnade eller ordar sig på ett annat sätt.
Strukturen hos enzym är inte statisk utan de olika delarna, aminosyrorna rör hela tiden på sig lite. Om strukturen destabiliseras (t.ex. genom ändrat pH, värme) försvinner en del stabiliserande bindningar och (delar av) strukturen börjar förlora sin struktur, t.ex. några helixar eller en domän. Och dessa förändringar sker på flera platser i strukturen samtidigt, oftast förändras strukturen så att enzymets aktivitet försämras - vissa delar rör kanske för mycket på sig, andra får en lite annan laddning.
Och dessa förändringar sker innan proteinet denaturerar helt när massvis med andra typer av nya bindningar bildas och många proteinmolekyler geggar ihop (motsvarande stekt ägg). Då är proteinet i ett tillstånd som inte längre kan vara löst i vatten och inte heller i t.ex. olja - utan det blir motsvarande en egen fastare fas.
Men skiftar pH från enzymets favorit pH och sedan tillbaka (t.ex. från pH 7 till pH 5 med låg aktivitet, tillbaka till pH 7 med optimal aktivitet) ändras laddningen på protonerbara sidokedjor - och de destabiliserade helixarna/domänerna kan då återfå en mer ordnade strukturen, och kan då katalysera reaktionen optimalt igen.
De övergripande förändringarna påverkar proteiner generellt. Det blir till slut en påverkan på bindandet av jonerna, men min gissning är att det är pH-orsakade förändringarna av proteinets struktur som får aktiviteten att gå ner - snarare än att jonerna inte kan binda in lika väl. (Det finns nog forskningsartiklar som beskriver detta, men dessa är inte så lättlästa, även för studenter på universitetsnivå).
Jag har även klurat på om det kan vara så att de kan påverkar den aktiva siten, dvs. blockera den genom att binda till någon negativ / positiv laddning som finns där och hindra stärkelsen från att binda. Ligger det något i det?
För pH 3 och 11 förstörs enzymt totalt, men för lite högre pH där hastigheten var mycket lägre, har det med att substansmängden icke-denaturerade enzym har minskat rejält, eller att det finns någon som gör de långsammare? Jag lutar åt det första, för jag tänker mig att antingen fungerar enzymet eller så gör det inte. Korrekt? Själv är jag osäker.
Det kan vara så att enzymet denaturerar helt vid pH under 3 eller över 11, och inte kan veckas rätt igen. Det är dock bra att komma ihåg att det du mätt är enzymets aktivitet, som försvinner vid dessa pH. Du vet med andra ord inte om enzymet är förstört, men du kan anta att det är denaturerat, och därför inaktivt - för proteiner brukar denaturera när pH förändras mycket. Lite petigt kan man tycka, men det är skillnad på om mörkret i rummet (ingen aktivitet) är för att lampan är trasig (denaturerat), eller om brorsan inte orkar resa sig från sängen och tända lampan (inte den aktiva strukturen, men inte heller stekt ägg).
3. Teori kopplad till just pH 6-7, det vill säga, exoenzymet anpassats efter förhållanden som bakterier trivs i, vilka ligger runt det pH:et, varför vet jag inte. Har det någon evolutionär orsak?
Absolut, under evolutionen har proteinerna i organismen anpassats för att fungera optimalt vid det pH som finns inuti organismens celler. Och lever bakterien kring neutralt pH är det fördelaktigt för organismen att anpassa sitt inre pH till detta "yttre" pH - för att på så vis slippa spendera massvis med energi och resurser på att "aktivt arbeta" för att hålla ett annat pH i cellen är den som finns precis utanför.
Så beroende på var denna bakterie brukar leva, har dessa amylas anpassats till att vara verksamt vid detta pH. Tänk även på att detta enzym är aktivt utanför cellen, i syfte att bryta ner stora stärkelsepolymerer till mindre sackarider, som cellen sedan kan ta upp. Precis som i vårt matsmältningssystem, så det finns en selektion för att få ett så pass effektivt enzym som möjligt - därför kan enzymets optimala pH vara olika från pH i våra celler (extremfallet i vår kropp är nog pepsin som bryter ner proteiner i magsäcken, med maximal aktivitet vid pH 2, jämfört med ett pH på ca 7 inuti de celler som bildar pepsinet).
För bakterierna i vår munhåla får indirekt nytta av att vi själva bildar amylas för att börja smälta maten. När vi processar maten, och även bryter ner matrester mellan tänderna (så det inte byggs upp och blir problem där) - frigör vi sackarider som bakterierna tar upp och använder i sin metabilism (som i sin tur sänker pH i munhålan).
Vi använde som du misstänkte bakteriellt amylas. De verkar inte kolonisera människor, så att salivens lägre pH verkar inte ha med det att göra som man annars kan tro för bakterier som lever i munhålan och använder amylas. Tillägg: kom just ihåg sura marker då bakterien lever i jord, kan det vara en evolutionär sak, alltså näringsfattig jord där även pH är lågt?
4. Felkällor kring okulär besiktning av färg, att det kanske ändå fanns viss mycket svag aktivitet runt pH 3 och 11 som inte kunda upptäckas, för visst kan man prata om aktivitet i detta sammanhang?
Ser framemot at höra vad du tycker!
Hälsningar.
Ja det blir en begränsning i metoden att du behöver bestämma "att nu har färgen förändrats", d.v.s. efter så här många minuter har stärkelsen brutits ner - vilket i sig är ett mått på aktiviteten. Det blir lite mer av ett subjektivt mått - "typ nu är det brunt" - men det fungerar det också, även om en spektrofotometrisk avläsning skulle ge ett mer objektivt siffervärde.
Det blir lite liknande "nu är korven färdiggrillad", du och jag har nog lite olika syn på när korven är färdiggrillad. Men grillar vi fem korvar på fem olika grillar med olika hetta, kommer vi se samma trend - det går väldigt fort i den varmaste grillen, och inget händer i den kalla, trenden kommer m.a.o. vara den samma oavsett om du gillar dina korvar lätt grillade eller svarta.
Jag har läst igenom tråden igen och undrar om du kan förtydliga ett fåtal saker:
Ja typ så, högst aktivitet såg du vid pH 6. Du kan även sätta detta intervall, mellan två pH värden, som en aktivitetsnivå - t.ex. högre än 30% av den maximala aktiviteten sågs inom pH 5-9, eller ännu smalare 50% av den maximala aktiviteten fanns mellan pH 5,5-7,5 i diagrammet. Då uttalar du dig inte om de data du saknar (t.ex. pH 5,5 och 6,5) utan om trenden i kurvan - som illustrerar hur pass pH känsligt enzymets aktivitet är.
Är detta fiktiva siffror? Hur kan man se ungefärlig andel genom att kolla på diagrammet? Det verkar som en smidig lösning att presentera resultatet på, men jag lyckas inte klura ut hur man kan räkna ut.
Hur kan jag styrka att denatureringen är irreversibel? Jag läste till mig att reversibla denatureringar är mycket ovanliga, men jag vill gärna backa upp mitt påstående i rapporten, har du några briljanta idéer?
Jag har funderat mer på felkällan; vad hade ett annat sätt varit? Jag tänkte på någon typ av färgmätare, men har det något annat proffsigare namn eller heter det bara färgmätare? Kan man använda spektrofotometri till detta? Alltså istället för jodprovet, mäta koncentrationen stärkelse efter x antal minuter? Jag har aldrig använt det så jag vet inte om det passar.
Angående mätosäkerhet, eftersom vi mätte med 15 s intervall och droppade enzymlösning i joden, kan det ju ha varit 15 s för mycket som vi stannade tiden på om stärkelsen redan brutits ner efter att reagensen ägt rum (förstår du vad jag menar?). Är mätosäkerhet 15 sekunder? Eller 7.5, dvs. hälften? Alltså den kan vara som mest 15 sekunder för mycket, men inte för lite (vad jag kan komma på), bortsätt från observationsaspekten.
Tack så mycket!
Mesopotamia skrev:Jag har läst igenom tråden igen och undrar om du kan förtydliga ett fåtal saker:
Ja typ så, högst aktivitet såg du vid pH 6. Du kan även sätta detta intervall, mellan två pH värden, som en aktivitetsnivå - t.ex. högre än 30% av den maximala aktiviteten sågs inom pH 5-9, eller ännu smalare 50% av den maximala aktiviteten fanns mellan pH 5,5-7,5 i diagrammet. Då uttalar du dig inte om de data du saknar (t.ex. pH 5,5 och 6,5) utan om trenden i kurvan - som illustrerar hur pass pH känsligt enzymets aktivitet är.
Är detta fiktiva siffror? Hur kan man se ungefärlig andel genom att kolla på diagrammet? Det verkar som en smidig lösning att presentera resultatet på, men jag lyckas inte klura ut hur man kan räkna ut.
Nja inte helt fiktiva, utan snarare en (kanske inte 100% exakt) avläsning ur diagrammet du visade. Du kan läsa ut mer exakta värden ur diagrammet, eller ange värdet lite mindre precist - med ett "ca 30% av den maximala aktiviteten". Jag uppskattade det till 30%, men du kan nog läsa ut det mer exakt ur diagrammet om du vill.
Och det går att räkna ut det exakt också, men det är verkligen overkill, och ger inte mer stöd till din slutsats - den kan bli densamma vid ca 30% som vid 33,6% (hittade på siffrorna, för att belysa att skillnaden till det "exakta värdet 33,6" inte är av intresse i sammanhanget - utan trenden är det, och den syns med ca 30%).
Hur kan jag styrka att denatureringen är irreversibel? Jag läste till mig att reversibla denatureringar är mycket ovanliga, men jag vill gärna backa upp mitt påstående i rapporten, har du några briljanta idéer?
Finns nog en risk att fastna i definitioner av vad denaturering är, reversibel, icke-reversibel, delvis reversibel. Du såg att aktiviteten inte fanns vid bl.a. pH 3, och du kan även påstå att det troligtvis beror på att enzymet är denaturerat. Mer än så behöver du inte säga, och du har inte riktigt resultat som styrker något annat.
Hade du haft tid kunde du kanske undersökt om avsaknaden av aktivitet berodde på att enzymer denaturerat på ett icke-reversibelt vis. Det kan kanske läggas till som ett "förslag på förbättring". Kommer du på hur det skulle kunna testas?
Jag har funderat mer på felkällan; vad hade ett annat sätt varit? Jag tänkte på någon typ av färgmätare, men har det något annat proffsigare namn eller heter det bara färgmätare? Kan man använda spektrofotometri till detta? Alltså istället för jodprovet, mäta koncentrationen stärkelse efter x antal minuter? Jag har aldrig använt det så jag vet inte om det passar.
Det ni såg var ett skifte i färgen på jodlösningen, när stärkelsen höll på att brytas ner. Denna förändring i färgernas intensitet (mindre lila och mer brun), kan absolut kvantifieras med en spektrofotometer.
Med risk för att det blir för mycket detaljer, så tittar ni efter det som kallas en aktromatisk punkt - när ingen färg uppfattas från jodlösningen. Motsvarande när ett färgomslag har skett, från det lila med jod i helikal stärkelse - till svagt brunt/ofärgat utan jodkomplexet i stärkelsen. Med en spektrofotometer kan man mäta när intensiteten för den lila färgen minskar och når "noll". Och Görs detta i realtid får man tiden till den akromatiska punkten kvantifierad med högre precision.
Men för en sådan laboration som ni gjort räcker det med att med ögat bestämma att "nu har stärkelsen brutits ner".
Angående mätosäkerhet, eftersom vi mätte med 15 s intervall och droppade enzymlösning i joden, kan det ju ha varit 15 s för mycket som vi stannade tiden på om stärkelsen redan brutits ner efter att reagensen ägt rum (förstår du vad jag menar?). Är mätosäkerhet 15 sekunder? Eller 7.5, dvs. hälften? Alltså den kan vara som mest 15 sekunder för mycket, men inte för lite (vad jag kan komma på), bortsätt från observationsaspekten.
Inte riktigt med på vad du menar. Ni startade flera parallella reaktioner, med 15 sekunders intervall kanske? Första vid t=0, andra vid t=15 s o.s.v. eller?
Och sedan observerade ni tiden det tog för proverna att avfärgas misstänker jag. Då bör ni ha ganska precisa tider, eller räknade ni även stopptiderna i 15 s intervaller?
Punkt 1: okej! Jag förstod dock inte hur man avläser alls i diagrammet... är det typ så:

Punkt 2: det jag kommer på är att man höjer pH igen med hjälp av en bas (eller buffert) och ser om aktiviteten ökar igen eller inte. På så sätt kan man veta om det beror på inaktivitet eller denaturering, kan det stämma?
Punkt 3: Tack för att du förklarade. Jag ska ta med det i förbättringen.
Punkt 4: Jag startade en timer så fort enzymet tillsatts till stärkelsen, och sen hällde jag en droppe på en joddroppe var 15 s fram till ingen färgskiftning på en ny joddroppe var märkbar, sen stannade jag tiden.
Det jag funderar på är att säg droppe nr. 10 som jag häller, när det har gått 150 s, så ser jag en liten förändring. Efter 15 s droppar jag nr. 11 och ser då ingen förändring. Men det jag antecknade ner var inkluderat den sista 15 s för droppe 11. Jag vet således inte om det egentligen var 5 s efter droppe 10 eller 10 s, jag vet bara att efter 15 s fanns ingen skillnad. Förstår du? Undrar dock vad det har för mätosäkerhet som jag kan skriva som felkälla. Inom fysiken brukar jag ta max-min/2 och få ett plusminus värde. Nu verkar det inte funka i sammanhanget, men det är tydligt är det är nån osäkerhetszon på 15 s.
Hoppas att det jag skriver går att någorlunda förstå, annars kan jag rita en bild!
Supertack!
Mesopotamia skrev:Punkt 1: okej! Jag förstod dock inte hur man avläser alls i diagrammet... är det typ så:
Du kan välja lite som du vill, så länge det tillför något.
Mitt förslag var något i stil med det nedan, där den maximala aktiviteten är vid den vita linjen, och det pH intervall där mer än halva maxhastigheten finns kan då fås genom den gröna linjen (halva maxaktiviteten) som finns inom det pH intervall som de blå linjerna avgränsar. D.v.s. mellan pH ca 5,4 och 7,8 har enzymet mer än halva maxhastigheten. Det var mest ett förslag för att visa dig att enzymernas aktivitet finns inom intervall av pH.
Det ger lite mer information för hur amylaset fungerar, än att bara presentera att "enzymet är mest aktivt vid pH 6". Information om detta intervall, och gränserna för när aktiviteten försvinner, tillsammans med maxaktiviteten, är relevanta för jämförelser i ett biokemiskt/biologiskt perspektiv. Ett annat amylas, eller annat enzym, kan också ha maximal aktivitet vid pH 6, men ett väldigt smalt intervall (pH 5,5-6,5) - och denna skillnad kan troligtvis förklaras utifrån organismens behov situation etcetera.

Punkt 2: det jag kommer på är att man höjer pH igen med hjälp av en bas (eller buffert) och ser om aktiviteten ökar igen eller inte. På så sätt kan man veta om det beror på inaktivitet eller denaturering, kan det stämma?
Ja det skulle man kunna göra. Återfås aktiviteten efter att pH justerats tillbaka till 6, så är det ju en reversibel effekt. Återfås väldigt lite eller ingen aktivitet efter justeringen, är det mer troligt att enzymet denaturerat helt vid t.ex. pH 3.
Punkt 3: Tack för att du förklarade. Jag ska ta med det i förbättringen.
Punkt 4: Jag startade en timer så fort enzymet tillsatts till stärkelsen, och sen hällde jag en droppe på en joddroppe var 15 s fram till ingen färgskiftning på en ny joddroppe var märkbar, sen stannade jag tiden.
Det jag funderar på är att säg droppe nr. 10 som jag häller, när det har gått 150 s, så ser jag en liten förändring. Efter 15 s droppar jag nr. 11 och ser då ingen förändring. Men det jag antecknade ner var inkluderat den sista 15 s för droppe 11. Jag vet således inte om det egentligen var 5 s efter droppe 10 eller 10 s, jag vet bara att efter 15 s fanns ingen skillnad. Förstår du? Undrar dock vad det har för mätosäkerhet som jag kan skriva som felkälla. Inom fysiken brukar jag ta max-min/2 och få ett plusminus värde. Nu verkar det inte funka i sammanhanget, men det är tydligt är det är nån osäkerhetszon på 15 s.
Ah ok, nu är jag också med.
Du har rätt att det blir en osäkerhet. Du saknar dock data för att veta om felet var 5 eller 10 s, som du skrev - så det blir svårt att på något vettigt vis ange detta. Det går så klart att räkna på det och ange ett "uppskattat fel på 1-15 sekunder" men det tillför inte något till tolkningen av resultaten, tycker i alla fall jag. Det blir precis samma fel på alla mätvärden, så det är nog bättre att ange mätonogranheten istället.
Istället kan jämföra och kommentera onogranheten med hur lång tid det tog tills det inte blev någon färg. 10 s är ganska mycket jämfört med en minut, men inte så mycket på 8 minuter.
Kan du komma på ett enkelt vis att öka nogranheten?
Hoppas att det jag skriver går att någorlunda förstå, annars kan jag rita en bild!
Supertack!
För all del, kul att se att du kämpar på!
Red: infogade bilden till slut


