Bioteknik, singlecell transcriptomics: exempel på heterogena cellpopulationer?
Hej, jag vill förstå motivationen bakom singlecell transcriptomics.
Jag fattar att om man inte har tillgång till detta så får man nöja sig med ett genomsnittligt transcriptom av en större population vilket kan maskera viktig information om individuella celler, men till en hur stor fysisk skala behöver vi zooma ut för att faktiskt kunna få en grupp av celler med unika transkriptom och som man därmed skulle ha intresse av att singlecellsekvensera?
Såhär skrev wikipedia:
These studies are limited as they provide measurements for whole tissues and, as a result, show an average expression profile for all the constituent cells. This has a couple of drawbacks. Firstly, different cell types within the same tissue can have distinct roles in multicellular organisms. They often form subpopulations with unique transcriptional profiles. Correlations in the gene expression of the subpopulations can often be missed due to the lack of subpopulation identification.[1] Secondly, bulk assays fail to recognize whether a change in the expression profile is due to a change in regulation or composition — for example if one cell type arises to dominate the population. Lastly, when your goal is to study cellular progression through differentiation, average expression profiles can only order cells by time rather than by developmental stage. Consequently, they cannot show trends in gene expression levels specific to certain stages.[4]
Men då skulle jag vilja ha några konkreta exempel på vilka vävnader detta kan handla om och varför de är intressanta?
men till en hur stor fysisk skala behöver vi zooma ut för att faktiskt kunna få en grupp av celler med unika transkriptom och som man därmed skulle ha intresse av att singlecellsekvensera?
I princip så små skalor att det finns två eller flera distinkta populationer av celler, där de respektive cellpopulationerna har sin egen typ av transkriptom (olika genuttryck m.a.o.). Men du kan även tänka dig att inom dessa populationer (t.ex. astrocyt vs glial cell), så kan det finnas en subpopulation av t.ex. astrocyter, vilka har en förhöjd transkription av vissa mRNA jämfört med resten av astrocytpopulationen.
Tas t.ex. en biopsi med nål/sprutspets, fås en väldig massa celler, och av olika celltyper, även nålens diameter är ganska liten (typ 1.6 mm). I den lilla biten av vävnad som fås ut ryms olika vävnadslager, även om det är en mer homogen vävnad som provet tas ifrån. Om en celltyp i denna vävnad är av intresse för diagnostik, t.ex. astrocyterna, så ökar så klart "signal to noise" för skillnader i transkriptom/expressionen, om singel cell transcriptomics används.
Det blir m.a.o. tydligare att det i just denna celltypen av intress finns onormalt höga nivåer av mRNA kodande för a,b,c,d, och normala nivåer av e-h. Med denna mer detaljerade information kan t.ex. en tumörcellpopulations utveckling följas. T.ex. behöver vissa förändringar i metabolismen ske för utvecklingen av en tumörcell (t.ex. a-d behöver öka). Har inte denna förändring i metabolism skett, är sannolikt inhibitorer som försämrar metabolismen (genom att försämra a-d) mer effektiva vid behandling, jämfört med om a-d redan har ökat. Eller a-d är mindre viktiga i metabolismen därför att e-h används betydligt mer, som en effekt av att metabolismen ändrats.
Fria celler, motsvarande de som cirkulerar runt i kroppen, har även de skillnader i genutrycket. Aktiveras ett fåtal minnes B-celler i immunförsvaret, som en effekt av att samma förkylningsvirus dyker upp igen, så kommer endast de aktiverade minnes B-cellerna att dela sig och bilda massvis med antikroppar. Så tittar du på transkriptom från B-celler generellt, skiljer detta markant ifrån subpopulationen "aktiverade minnes B-celler".
Ok! Alltid så tydliga och uttömmande svar, tack så mycket!
För all del, kul med lite spetsiga frågor också :)
Detsamma, så bra att du kan så mycket!
Men ett litet problem jag ser (kan du se om det stämmer?) är att informationen om cellernas spatiella relation antagligen försvinner vid SCS (singecell sequencing), eller hur? Om man använder tex cell sorting som i FACS.
Det har inte så stor betydelse för fria celler som ändå beblandas, men tex i cancersammanhang?
På wikipedia nämner de fem sätt att separera celler:
Micropipetting
Cytoplasmic aspiration
Laser capture microdissection.
Samt high-throughput metoder:
- Fluorescence activated cell sorting (FACS)
- Microfluidic devices
Jag vet inte så mycket om dessa men skulle laser capture kunna bevara spatiell info?
En annan fråga:
Det står på wiki att långsamma metoder kan isolera några hundra och high thorughput ett antal tusen, är det ens mycket i sammanhanget? Är det inte önskvärt att ha (mycket) mer?
Tas t.ex. en biopsi med nål/sprutspets, fås en väldig massa celler, och av olika celltyper...
Hur mycket är detta ungefär? Om vi utgår från en cellpopulation som inte är fritt blandat (blodceller) kan det inte hända att sjuksköterskan sticker lite snett och råkar få exakt en homogen population?
Qetsiyah skrev:Men ett litet problem jag ser (kan du se om det stämmer?) är att informationen om cellernas spatiella relation antagligen försvinner vid SCS (singecell sequencing), eller hur? Om man använder tex cell sorting som i FACS.
Det har inte så stor betydelse för fria celler som ändå beblandas, men tex i cancersammanhang?
På wikipedia nämner de fem sätt att separera celler:
Micropipetting
Cytoplasmic aspiration
Laser capture microdissection.
Samt high-throughput metoder:
- Fluorescence activated cell sorting (FACS)
- Microfluidic devices
Jag vet inte så mycket om dessa men skulle laser capture kunna bevara spatiell info?
Så länge du kan isolera en begränsad mängd celler, och relatera det till sin position i objektet (t.ex. en av 100 subvolymer av ett vävnadsprov) så borde den spatiella upplösningen kunna bibehållas.
Jo med laser capture microdissection kan du bibehålla en spatiell upplösning. Men det brukar ske ifrån tvådimensionella preparat, motsvarande mircotome-preparat på objektglas, vilket borde fungera (så länge RNAse och andra mRNA nedbrytande processer hämmas). Har inte koll på hur mycket energi som deponeras i samband med metoden, och hur stor skadan då kan bli på preparatets celler. Känns inte riktigt som om metoden är mogen för icke-destruktiv isolering av t.ex. celler. Men vill du kolla på endast en del av ett tunt skikt, kan t.ex. fettceller "lyftas" bort från omkringliggande kapillärer etcetera. Det är en rätt häftig metod för att från ett prov "lyfta" och därmed isolera väldigt begränsade delar, som sedan kan studeras/analyseras individuellt.
Qetsiyah skrev:En annan fråga:
Det står på wiki att långsamma metoder kan isolera några hundra och high thorughput ett antal tusen, är det ens mycket i sammanhanget? Är det inte önskvärt att ha (mycket) mer?
Beror nog helt på frågeställning. Transkriptionsnivån på några hundra gener tror jag kvalitativt ger rätt mycket information, om du analyserar t.ex. onkogener, gener för metabolism, cell-cykel regulatoriska gener, så får massvis med information om tumörcellens "fenotyp".
Tas t.ex. en biopsi med nål/sprutspets, fås en väldig massa celler, och av olika celltyper...
Hur mycket är detta ungefär? Om vi utgår från en cellpopulation som inte är fritt blandat (blodceller) kan det inte hända att sjuksköterskan sticker lite snett och råkar få exakt en homogen population?
Jo det borde väl gå, om objektet är homogent. Men för att en tumör skall kunna växa till sig krävs finns förutom tumörcellerna även vaskularisering, d.v.s. någon form av (blod)kärl, som förser tumören med syre/näring och tar med sig restprodukterna. Så en helt homogen population av tumörceller är nog svårt att få, men det kanske inte är ett måste, för även om "andra" celler följer med och skapar brus i provets transkriptom, så innehåller referensvärdena nog en del brustranscripts.
Tillägg: 15 nov 2022 23:07
En sak till som jag missade att skriva. Det finns så klart en gräns för hur små volymer som en biopsi kan tas ifrån. Är den misstänkta tumören liten, blir det svårt om inte omöjligt att med hyfsad säkerhet träffa tumörvolymen och få en "bra" biopsi, som innehåller tumörceller och inte andra celler runt omkring den misstänkta tumören. Har dock ingen siffra för volym att ge, men en "felaktigt" tagen biopsi, med friska celler i, har ett negativt/missvisande värde i samband med diagnostik.
Okej!! Tack återigen
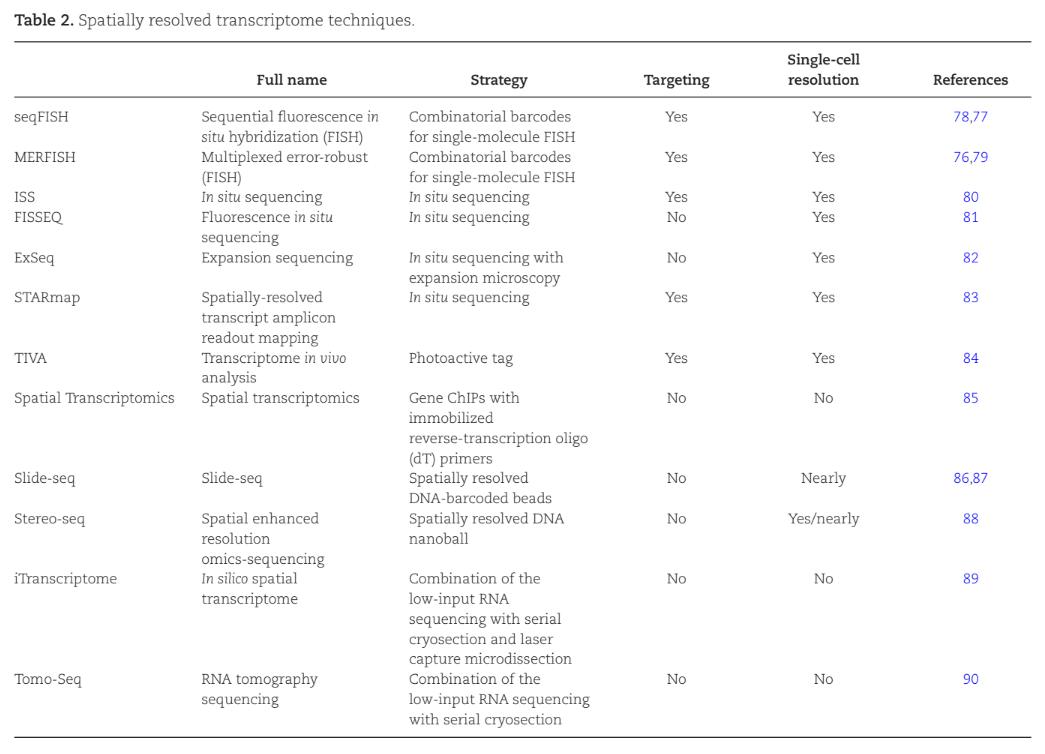
Hej igen, kan du förklara hur en teknik kan ha spatiell upplösning, men inte cellulär upplösning? (motsatt fråga till hur en teknik kan ha cellulär men inte spatiell). Betyder det helt enkelt bara att upplösningen inte är tillräckligt bra?
Jag läser https://pubmed.ncbi.nlm.nih.gov/36254761/ och där finns bilden
Jag försöker bilda mig en uppfattning om vad som pågår inom det senaste och hetaste, men det är så svårt när artiklar för bara >3 år sen kan bli inaktuella...
Den spatiella upplösningen är väl främst kopplad till cellens relation till volymen cellen kom ifrån. Motsvarande x,y,z koordinater i en volym av en vävnad. Går det inte att erhålla individuella celler under provberedningen och samtidigt bibehålla cellens position, saknas den allra högsta spatiella upplösningen, RNA-nivåer från en enskild cell med en definierad position i ett koordinatsystem.
Eller som i tabellen, där gen chip inte klassas som single cell resolution, troligtvis för att själva chippet innehåller snuttar av gensekvenser "probes" och urskiljningslöst fångar komplementärt DNA. Och det upparbetade cDNA som används vid hybridiseringen till dessa probes, behöver inte nödvändigtvis komma ifrån individuella celler med en bestämd koordinat.