Biokemi: bestämma jämviktskonstant för enzymkatalyserad reaktion
Hej, se denna uppgift:

Och den givna "datan":

Men jag är helt borta, på föregående del av labben skulle vi ta reda på reaktionshastighet m.h.a. en hjälpreaktion och spektrofotometer. Vi tillsatte enzymet sist och la kyvetten i spkr så snabbt som möjligt för att se den initiella reaktionshastigheten, men nu vill vi att reaktionen ska nå jämvikt först, vad ska vi detektera ens då? jag fattar inte hela meningen med labben.
Och konkreta frågor:
- varför säger de inte våglängden som abs mäts vid? Jag antar att det är 340 (där NADPH absorberar).
- mätningen där abs=0,56, har de väntat en timme som det står i instruktionerna eller gjordes mätningen direkt efter tillsats av GPI?
Det är lite samma här, för det verkar som det enda viset du kan detektera något är genom NADP+, som kommer reduceras till NADPH, när GPDH oxiderar G6P till 6-fosfogluco-delta-laktonen. Och vatten kommer attackera och konsumera denna, för att bilda karboxylsyran 6-fosfoglukonat.
Isomerasets jämvikt bör med råge ha infunnit sig efter en timme, och om du sedan tillsätter en större mängd av GPDH kommer NADP+ reduceras direkt, och absorbansökningen vid NADPH absorbansmaxima (eller reduktionen av NADP+ absorbansen) kommer vara direkt proportionell till mängden oxiderad G6P. Det enda kruxet är aktiviteten hos GPI, då jämvikten förskjuts - men om du har en väldigt låg koncentration av GPI (och längre tid för att jämvikten skall inställa sig) behöver du bara ha ett ordentligt överskott av NADP+ och GPDH för att snabbt som ögat oxidera bort allt bildat G6P - innan GPI kommit igång för att försöka etablera jämvikten igen.
På så vis kan du spektrofotometriskt kvantifiera hur mycket NADPH som bildas, och då vet du hur mycket G6P som fanns - och då du vet den ursprungliga substansmängden av F6P (du verkar kunna ange detta själv) kan du helt enkelt räkna ut hur mycket F6P som genom GPI konverterats till G6P.
Okej men vänta lite, så GPI ställer in sig först och ingen GPDH finns från början. (NADP+ finns från början men det spelar ingen roll om den gör det eller inte). Sedan tillsätts GPDH i extremt överskott för att bilda NADPH i 1:1 förhållande till G6P, och vi antar att GPI under tiden inte hinner skapa mer G6P.
Eftersom stökiometrin är enkel så blir jämviktskonstanten kvoten [F6P]/[G6P]. [F6P] vet jag från början, jag bestämmer den själv. [G6P] får jag genom lambert beers lag och absorbansen direkt efter GPDH?
Men i infon får jag två experimentella data, har jag ingen användning av abs=0,46 isåfall?
Qetsiyah skrev:Okej men vänta lite, så GPI ställer in sig först och ingen GPDH finns från början. (NADP+ finns från början men det spelar ingen roll om den gör det eller inte). Sedan tillsätts GPDH i extremt överskott för att bilda NADPH i 1:1 förhållande till G6P, och vi antar att GPI under tiden inte hinner skapa mer G6P.
Precis, det skulle vara ett sätt att detektera koncentrationen/substansmängden av G6P efter att GPI ställt in jämvikten till F6P. Som jag ser det kan du inte kvantifiera någon annan koncentration än den av NADP+ (alternativt NADPH beroende på våglängd, men se senare kommentar om detta).
Eftersom stökiometrin är enkel så blir jämviktskonstanten kvoten mellan: uppmätt abs direkt efter tillsats av GPDH och... vad? Vad är abs=0,46 egentligen?
Precis, det finns endast två koncentrationer att ta med i beräkningen, den ena bestämmer du spektrofotometriskt och den andra vet du redan från början.
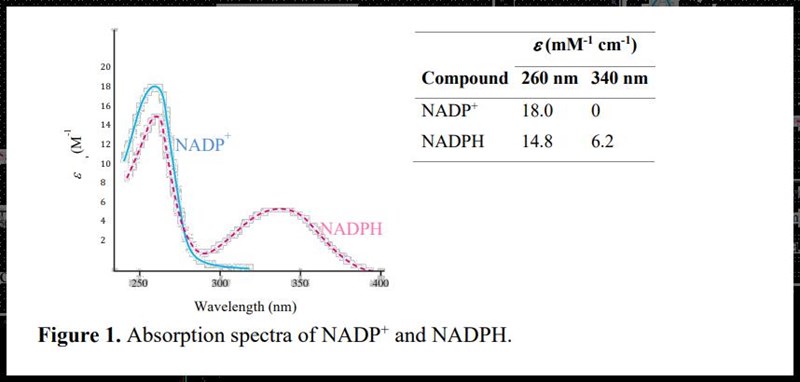
Gällande absorbansen, tja det beror på våglängden. Men då detta är dina experimentella data misstänker jag att detta är den våglängden där NADPH har en egen topp i spektrumet ~340mn. Denna absorbans är resultatet av G6P-reduktionen.

Så vad behöver du ha som nollprov?
Ah sorry, absorbansen minskade - då bör det vara NADP+ toppen vid 225 nm.
Nej vänta jag skrev fel. Jag menar abs=0,56, vad har jag för använding för den? Jag förstår inte vilken data som är vilken, men hursomhelst finns det finns det två data, och det är bara en av de vi har konstaterat att jag behöver.
Varför får jag datan i denna konstiga ordning? GPI tillsätts först, sedan GPDH?
Vi har också en sån bild:
Asså det vi vill nolla bort är väl absrobansen hos enzymerna i sig?
Detekterar du vid 260 nm kommer de aromatiska aminosyraresterna bidra till absorbansen. Det verkar nästa som detta är fallet då abs är 0,56 AU för "100µl GPI (från stock lösning)", för som jag tolkar det tillsätts NADP+ först senare och tillsammans med GPDH.
Men då NADP+ absorberar friskt, och tillsätts samtidigt som det direkt förbrukas, behöver du kunna korrigerar för det som tillsätts till jämviktsblandningen - annars har du ingen referensabsorbans att utgå ifrån.
Men mina kompisar säger att båda mätningar är vid 340, vad tycker du att mätningarna är tagna på för längd?
Det klassiska sättet är så klart att mäta absorbansökningen för NADPH vid 340nm.
Men jag fastnade vid att kyvetten endast innehållandes GPI har en så hög absorbans som 0,56 AU. GPI saknar kofaktorer eller annat som absorberar vid 340 nm. Det finns säkert något (exotiskt) protein utan kofaktorer som absorberar lite vid 340 nm, men proteiner brukar inte alls absorbera alls vid 340 nm (några kofaktorer gör det men inte proteinet i sig). Så att ett protein absorberar så mycket vid 340nm stämmer inte. Visst det går att kanske att hävda att t.ex. bufferten absorberar vid 340 nm, men det är en faktor som direkt skulle ha elimineras, helt enkelt genom att byta buffer - och då borde absorbansen för GPI lösningen vid 340nm vara noll - då detta enzym saknar kofaktorer.
Det beror lite på hur klurig uppgiftsmakaren har varit. Har hen varit lite klurig, är svaret att absorbansen mäts vid 260 nm. Men detta kan också vara en uppgift gjord "i undervisningssyfte" och då är mitt resonemang övertänkt. Det mest uppenbara är att mäta vid 340 nm vilket ger renare data, abs. för NADPH går från noll till 0,46 AU. Detta är trots allt en labb, och inte ett experimentet.
Dock stämmer inte GPI absorbansen vid 340nm. Vid 260 nm absorberar de aromatiska sidokedjorna i GPI och det vore inte orimligt att 0,56 AU skulle kunna fås från en stocklösning av detta enzym.
Är det vid 260nm som absorbansen mäts, kan du dels få en uppfattning om hur hög GPI koncentrationen (proportionell till abs) är samt räkna ut hur stort bidrag GPI ger till den totala absorbansen i provkyvetten (med allt i) - då du vet hur mycket du tillsätter (X µl med 0,56 AU spätt till 2 ml...). På så vis kan du "räkna bort" absorbansbidraget från GPI.
För det vore underligt om ni fick ett absorbansvärde för endast GPI, som ni inte skulle ha någon nytta av annars.
Okej, men är du säker på vad proven innehåller ens? Jag har inte förstått det än. Abs=0.56 innehålller ingenting annat än buffer och isomeraset? Och abs=0,46 är den vi pratar om ovan, alltså den där GPDH tillsätts i mycket stort överskott och som berättar om hur stor mängd G6P som fanns vid jämviktsläget?
Tja det är inte för tydligt skrivet, speciellt inte kopplingen mellan laborationshandledningen på engelska och "jämvikt uppfigt B" på svenska.
0,56 AU är för GPI stock-lösningen, med endast GPI (kanske med buffer, det brukar ju vara med, men måste inte vara med). Detta är 100µl och det stod i den engelska texten att ni skulle använda 2 ml, så detta är kanske spätt.
Sedan är det jämviktsblandningen x µl (bestående av 2mM F6P, GPI, efter 1 timmes inkubation), sedan tillsätts GPDH och NADP+. Och jag tolkar det som att abs på 0,46 AU är efter tillsatsen av allt till kyvetten (jämviktsbl., GPDH och NADP+) - d.v.s. detta är datat för den kopplade GPDH reaktionen.
För så snart som GPDH och NADP+ tillsätts sker oxidationen av G6P och NADPH bildas. Så GPI måste ju i först tillsammans med F6P för att bygga upp jämvikten mellan F6P och G6P, innan GDPH och NADP+ tillsätts.
Lite underligt dock att ni fått ett absorbansvärde på 0,46, men det blir nog lite struligt med olika abs. värden (som kommer ifrån de olika volymerna/koncentrationerna av de tillsatta lösningarna x,y,z).
Jaha det är det jämviktsblanding betydde, vad dum jag är.
Vi har båda missuppfattat. Jag frågade kompisar igen och det är som såhär: den första datan är tagen ungefär som du föklarade ovan, men för att vara på säkra sidan har GPI:n blivit denaturerad (med värme) innan tillsats av DGPDH. Sedan tillsätts MER GPI för att ta reda på hur mycket F6P som fanns i jämviktsblandningen.
Således är den första abs proportionell mot konc G6P och den andra proportionell mot konc G6P. Lambert beers lag behöver väl då inte ens användas? (volymen ny GPI lösning är försumbar?) Det är bara att dela absorbanserna.
Eller vänta, visst behöver jag ta skillnaden av absorbansera? NADPH som kom av G6P är ju fortfarande kvar.
Qetsiyah skrev:Jaha det är det jämviktsblanding betydde, vad dum jag är.
Vi har båda missuppfattat. Jag frågade kompisar igen och det är som såhär: den första datan är tagen ungefär som du föklarade ovan, men för att vara på säkra sidan har GPI:n blivit denaturerad (med värme) innan tillsats av DGPDH. Sedan tillsätts MER GPI för att ta reda på hur mycket F6P som fanns i jämviktsblandningen.
Det saknas en del information i texten (men det kanske är lite av utmaningen med uppgiften, eller så har ingen orkat fixa till den). Denatureringen förhindrar vidare GPI omvandling, så att all bildad G6P kan oxideras, vilket ger mer tid för det andra enzymatiska steget.
Och att efter att all bildad G6P oxiderats tillsätta ytterligare GPI för att omvandla kvarvarande F6P går så klart. Men GPDH reaktionen är reversibel och då det tar tid för omvandlingen av kvarvarande F6P med GPI kommer experimentet i kyvetten ta mer tid, och jämvikten mellan G6P <=> 6-phospho-D-glucono-1,5-lactone hinner ställa in sig. Denna jämvikt kommer vara förskjuten mot 6-phospho-D-glucono-1,5-lactone (då NADP+ är i 5 ggr. överskott över F6P) - men det blir ytterligare en jämvikt att ta hänsyn till. Dessutom kommer koncentrationen av G6P som bildas med den ytterligare GPI sannolikt vara hastighetsbegränsande för GPDH steget, och då kommer [G6P] hela tiden vara låg och [laktonen] öka - vilket ökar hastigheten för reduktionen av 6-phospho-D-glucono-1,5-lactone tillbaka till G6P. Så denna GPDH jämvikt blir knepigare att bestämma på ett exakt vis. Skall det bli korrekt behöver sista stegets jämvikt elimineras, idealfallet skulle sista GPDH steget vara irreversibelt, om reaktionen skall pågå under en längre tid.
Detta är nog som sagt inte ett så stort problem (på en studentlabb kanske det till och med får bli att bortse ifrån den andra jämvikten), men är inte ett korrekt sätta att mäta resterande [F6P]. Den extra tiden det tar GPI att konvertera F6P till G6P kommer att göra att GPDH jämvikten kommer att ställa in sig. På så vis kommer du inte att få 100% oxidation av G6P utan en del kommer vara kvar som G6P och F6P, så summan av dina [NADPH]med denaturerat GPI + [NADPH]med ytterligare GPI kommer aldrig bli samma som koncentrationen av F6P du började med när jämviktsblandningen preparerades.
Du har fördelen att du vet hur mycket F6P du börjar med, så du behöver egentligen endast bestämma koncentrationen av bildad G6P i jämviktsblandningen, differensen är den kvarvarande mängden F6P. Problemet med den andra jämvikten kvarstår dock och kan (nog) inte elimineras med de parametrar som finns i uppgiften - den andra jämvikten kvarstår och gör att [G6P] koncentrationen kommer underskattas.
Således är den första abs proportionell mot konc G6P och den andra proportionell mot konc G6P. Lambert beers lag behöver väl då inte ens användas? (volymen ny GPI lösning är försumbar?) Det är bara att dela absorbanserna.
Ja absorbansen bör vara proportionell mot G6P (genom NADPH), men all tillsatt lösning ökar ju volymen, så jag skulle inte alls säga att den är försumbar. Är volymen dessutom de 0,1 ml som är angivna i den andra lösningen blir felet enormt, speciellt när din datapunkt är koncentrationen.


