Dubbel - Överkorsningar vid homologa kromosomer.
Jag läser en grundkurs i genetik på högskola just nu. Känner mig rätt stabil med det mesta förutom vissa specifika områden.. Just när det kommer till Gen - mapping och linkage är jag hyfsat säker, då jag klarat de flesta instuderingsfrågor utan större problematik.
Men just dessa två får mitt huvud att ryka! Det jag vill ha hjälp med främst är fråga (1c) och hela fråga 2, gärna med förklaring då jag ej kan hitta något att relatera till varken i vår kurslitteratur eller föreläsning (mycket möjligt där är liknande exempel, men förstår man ej grunden är det svårt..) Fråga (1a-b) etc. får gärna avstämmas, men de känner jag mig mer säker på! Stort tack på förhand!
Med vänliga hälsningar, Azadriel
P.S, skriver på engelska då kursen ges enbart på just engelska.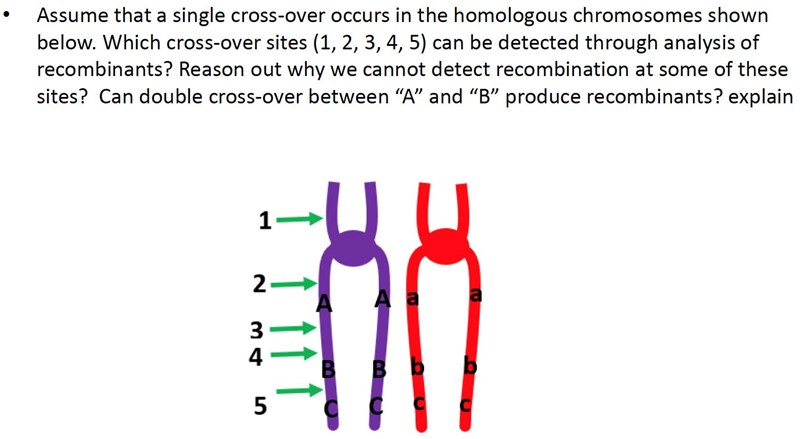
Svar fråga 1:
a) 3, 4 & 5. Since these sites are located between the marker genes.
b) At site 1 & 2, recombinants cannot be detected since they are outside the marker area. These sites are non - recombinant.
c) This is the question i'm uncertain of. Point 3 & 4 can create 2/4 recombinants? I don't feel too familiar with double crossing overs. Not explained too well at lectures either.. But could it be cause of more then two strands overlapping for example, Three strand which generates 2 parental and 2 recombinants? (Väldigt osäker på mina svar här)
(Väldigt osäker på mina svar här)
Fråga 2:
a) No clue about if a parental gamete can be produced.. My guess would however be no, since F & G is outside the recombinant region?
b) FGh, Fgh, fGH, fgH, fGh and fgh.
Oj, den här måste en biolog svara på, vilket det råder brist på här på pluggakuten, men en användare vid namn SvanteR kan hjälpa dig.
Välkommen
Qetsiyah skrev:Oj, den här måste en biolog svara på, vilket det råder brist på här på pluggakuten, men en användare vid namn SvanteR kan hjälpa dig.
Välkommen
Aha okej! Kan man tagga någon här? Eller kanske skicka Pm? tusen tack för snabb respons oavsett! Klistrar en sista följdfråga samtidigt ifall någon skulle svara.
3. Assume that six additive genes control a complex trait in diploid crop species and each gene has two alleles. Each uppercase allele is favorable for the expression of the trait and adds one unit to the phenotype. Each lowercase allele has no effect on the phenotype. For example:
- In the case of genotype, AABBCCDDEEFF, all alleles contribute to the phenotype and thus the plant has maximum value for the trait.
- In the case of genotype, aabbccddeeff, none of the alleles contributes to the phenotype and thus the plant has minimum (zero) value for the trait.
P1 x P2 = F1
AABBCCDDEEFF x aabbccddeeff = AaBbCcDcEeFf
F1 x F1 = F2
AaBbCcDcEeFf x AaBbCcDcEeFf
How many phenotypic classes do you expect at F2? And in what proportions?
9:3:3:1
9/16 x 14 400
3/16 x 14 400
3/16 x 14 400
1/16 x 14 400
En klasskamrat skickade detta till mig och har nu gått under jorden verkar det som.. Skulle någon kunna förklara detta för mig? Förstår ej hur man kommat fram till denna slutsats eller vad den ens har med frågan att göra..
Med vänliga hälsningar, Azadriel
Detta är inte direkt den genetik jag har färskast i minnet, men jag ska försöka skriva ett svar. Det kan dock dröja till i morgon!
Eller kanske skicka Pm?
Det kan man, fast det är inte tillåtet enligt forumreglerna. Vissa användare gillar det och andra gör det inte.
OK, först en liten reservation: Jag är ganska rostig, så ta mina svar med en liten nypa salt. Men jag tror jag kan svara och tar en fråga i taget:
"Can a double cross-over between A and B produce recombinants?"
En dubbel överkorsning mellan A och B betyder ju först en vid site 3 och sedan en vid site 4 (eller omvänd ordning, men det spelar ingen roll). Den första överkorsningen (vid site 3) gör ju så att den lila kromosom blir röd från site 3 och nedåt (och tvärtom för den lila). Nästa överkorsning (vid site 4) flyttar ju tillbaka en röd bit, så den blir röd från 4 och ned. Man får alltså en lila kromosom med en röd bit mellan 3 och 4. Men där finns ju ingen av generna A, B och C, så vi kan inte detektera den biten.
Sedan fråga 2: Varför skulle man inte kunna få en ickerekombinant gamet? Det är väl alltid det man får om det inte sker någon överkorsning! Så jag skulle svara ja på detta, om det inte finns någon fälla som jag missar.
När det gäller de möjliga rekombinanta gameterna så måste du komma ihåg att det står "assume that a single cross-over can occur". Du får alltså klippa och klistra men bara en gång! Till exempel fGh som du skriver kräver två klipp. Fundera igen på vilka som kan bildas genom att du kapar på ett ställe.
Till sist fråga 3: Här tolkar jag frågan som att det inte handlar om klassisk Mendelsk nedärvning med dominanta och recessiva gener (där A är dominant och a är recessiv och AA har samma fenotyp som Aa).
I stället verkar det vara fråga om en additiv effekt, där AA ger dubbelt så stor effekt som Aa. Då får man alltså maximal effekt om man har genotypen AABBCCDDEEFF. Eftersom det är 12 alleler får man 12 enheters effekt. Alla alleler har samma effekt, dvs en individ med AABBCCDDEeff ser likadan ut som en individ med aABBcCdDEEFF. (Jag tror inte att din klasskamrat har uppfattat frågan så, men baserat på det du lagt upp tror jag att hans svar är fel.)
Det betyder att man kan se hur stor effekten blir genom att räkna antalet stora bokstäver. Maximala möjliga antalet är 12 och det minimala antalet är 0, vilket betyder att det finns totalt 13 möjligheter. Svaret på antalet fenotypiska klasser blir alltså 13.
Vi antar att generna inte är länkade. Det betyder att hos F2-avkomman är alla kombinationer av alleler lika sannolika (eftersom F1 är heterozygot för alla gener). Genotypen AABBCCDDEEFF är lika sannolik som AAbbccDDeEFF eller vilken annan kombination som helst.
Men alla fenotyper kommer inte att vara lika vanliga. Det finns bara en enda genotyp som ger maximal effekt (AABBCCDDEEFF), men det finns 12 olika genotyper som ger den näst största effekten (aABBCCDDEEFF, AaBBCCDDEEFF, AAbBCCDDEEFF och så vidare). Det betyder att proportionerna mellan individer med störst effekt och individer med näst störst effekt kan skrivas 1:12.
För att få veta proportionerna får man friska upp den matte man lärde sig i Ma5, eller någon inledande kurs på universitetsnivå. Repetition finns här:
https://www.matteboken.se/lektioner/matte-5/kombinatorik/kombinationer
Genotyperna som ger den tredje största effekten är de där två stora bokstäver är utbytta mot små (till exempel AaBBCCDdEEFF). Hur många sådana finns det? Det beräknar man genom att räkna ut
Proportionerna för de tre klasserna med störst effekt blir då 1:12:66. Det betyder att för varje avkomma i F2 med störst effekt kommer det att finnas 12 med näst störst effekt och 66 med tredje störst effekt.
För att fortsätta beräkna proportionerna beräknar man , och så vidare. Om man vill slippa göra beräkningarna letar man upp en bild på Pascals triangel och skriver av värdena därifrån. Här finns en tillräckligt stor bild:
https://www.slideshare.net/vbhunt/pascals-triangle-31417851
Du får alltså läsa av raden som börjar 1:12:66:220:495 och fortsätta hela vägen.
Jag hoppas att detta svarar på din fråga. Som sagt så är jag lite rostig och kan ha missförstått frågan, så får du facit får du gärna återkomma. Detsamma gäller förstås om du undrar något mer!



